 Dominancia incompleta
Dominancia incompletaLa dominancia incompleta es una condición en la cual ningún alelo es dominante sobre el otro. La condición es reconocida para heterocigotas que expresan un fenotipo intermedio en relación a los fenotipos paternos. Si una planta roja se cruza con una planta de flores blancas, la progenie será toda rosa. Cuando una rosa se cruza con otra rosa, la descendencia es 1 roja, 2 rosas, y una blanca.
GENÉTICA HUMANA
Es e estudio de las ciencia de las variaciones humanas.
- Genoma : Es la totalidad de la información genética humana contenida en el ADN de las celulas de un individuo.
- Bianformatica : Es una rama de la biología que estudia loas secuencias de las bases del ADn humano y comprara con otras especies.
- Pedigrí (Árbol genialógico): Estudio ordenado de una o dos generaciones de una familia basandose en la presencia de caracteristicas siguiendo unos simbolos determinados.
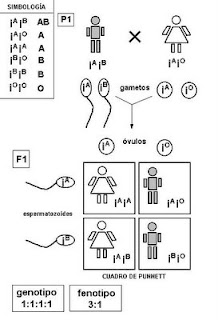 Codominancia
CodominanciaCuando la acción de los dos alelos presentes en el heterocigoto se manifiesta simultáneamente se dice que existe codominancia. Los alelos que se manifiestan simultáneamente en el heterocigoto reciben el nombre de codominantes.
Alelos Múltiples
Hablamos de alelos múltiples cuando hay más de dos alelos alternativos posibles para especificar ciertos rasgos.
Un ejemplo típico lo constituyen los alelos del sistema de grupos sanguíneos. Los alelos múltiples se originan de distintas mutaciones en un mismo gen.
Los 4 grupos sanguíneos: A, B, AB y O son resultado de tres diferentes alelos de un sólo gen (iA, iB e iO), iA e iB son codominantes sobre iO que es recesivo.
Los alelos iA e iB producen diferentes glucoproteínas (antígenos) en la superficie de cada eritrocito.
Los homocigotos para A producen el antígeno A, los de B sólo los del B, los de O, ninguno.
Sin embargo, los alelos iAiB son codominantes uno con el otro, es decir, ambos son fenotípicamente detectables en los heterocigotos.
Los individuos iAiB tienen eritrocitos tanto con glucoproteínas A como B y tienen sangre tipo AB.
CARIOTIPO
Conjunto de cromosomas de un organismo, los organismos y las plantas son diplodes tienen dos copias equivalentes de cada cromosoma.Siempre hay un proceso de pelicacion de la información, tenemos 23 pares de cormosomas.
CITOGÉNETICA
Estudio del cariotipo humano, Descripción y metodología .
CITOGENETICA HUMANA:
Estudia el conjunto de cromosomas de un individuo CROMOSOMAS METAFÍSICOS los cuales son cromosomas formados por dos cromatidas hermanas = dos moléculas de ADN idénticas asociadas a proteínas, eso ocurre en metafase.
- Metacentrico: cromosomas estan en la mitad y lo brazos son iguales.
- submetracentrico: brazo corto y uno largo.
- acrocentrico: cuando no tiene brazos si no satelites.
Bandas Q: Tratamiento con quinocrina ( busca regiones encargadas de adenina y timina)
Bandas G= Tratamineto en tripsina + gliemsa regiones ricas en AT ( bandas oscuras)
Bnadas R: Son inversas a G y Q y esta ricas en C G ( bandas oscuras)
Bandas C: Heterocrimatina constitutiva
REPLICACIÓN DEL ADN
Procedimiento necesario para que se realice la división celular. Ocurre en la fase S del ciclo celular, el mecanismo de repliación se basa en la complementación de bases. Inicialmente se plantearon tres posibles modelos de replicación :
- modelo conservativo: Mi ADN y lo copiaba.
- modelo dispersivo: Entraba ADN la copia cogía una hebra.
- modelo semiconservativo : hebra de ADN se abre y empieza una copia.
Transducción :
Se caracteriza porque en ella se puede transferir cualquier trozo de genóforo bacteriano, con tal de que tenga un tamaño compatible con la capacidad de “empaquetado” de ADN de la cápsida del fago. La partícula transductora (pseudovirión) se forma por empaquetamiento anómalo de ADN genofórico bacteriano. En el interior de la cabeza del pseudovirión sólo existe ADN bacteriano, sin ADN del fago. Las partículas transductoras sólo se forman como consecuencia de infecciones líticas del fago.
Mecanismo de la transducción generalizada promovida por el fago P22 de S. typhimurium.
1ª fase: producción de las partículas fágicas transductoras (=pseudoviriones): Por encapsidamiento ilegítimo de ADN cromosómico. De vez en cuando, el sistema fágico encargado de introducir su ADN en la cápsida (sistema pac), se “equivoca”, y en su lugar introduce un trozo de tamaño equivalente del genóforo de la bacteria donde está teniendo lugar la infección.
Al parecer, el cromosoma bacteriano contiene secuencias que eventualmente pueden ser reconocidas de vez en cuando por el sistema del fago, de manera que puede introducirse un trozo de ADN de Salmonella en la cápsida.
Al final de esta fase, la célula hospedadora se lisa. El lisado (sobrenadante) contiene una mayoría de viriones auténticos y un pequeño número de pseudoviriones, cada uno con un trozo aleatorio distinto del genomio de la bacteria.
2ª fase: Destino del ADN del exogenote: Mezclemos el lisado obtenido en la fase anterior con un cultivo de la cepa receptora (dotada de marcadores genéticos adecuados). Cada pseudovirión inyecta de forma normal su ADN a una bacteria. Este ADN puede tener varios destinos posibles:
a) puede ser destruido por exo- y endonucleasas citoplásmicas.
b) Puede recombinarse con la región homóloga del genóforo del receptor, mediante la actuación del sistema de recombinación general dependiente de RecA. Se produce una recombinación con dos sobrecruzamientos (“crossing-overs”), que conduce a la integración de doble cadena de ese exogenote, con eliminación de la zona homóloga del endogenote.
c) Puede ocurrir que el exogenote no sea ni destruido ni recombinado; este ADN puede persistir en la célula sin replicarse. La consecuencia de esto es que cuando la célula que originalmente recibió ese ADN exógeno se divida, lo pasará solo a una de las dos células hijas, la cual a su vez la pasará a una hija en la siguiente división, y así sucesivamente. Tenemos, pues, que el exogenote se va diluyendo en el clon por transmisión unilinear: tras varias generaciones, el clon consta de n-1 células sin exogenote y una sola célula con ese exogenote. A este fenómeno se le conoce con el nombre de transducción abortiva, y es más frecuente que la transducción completa derivada de recombinación (más de 90% frente a sólo 1-5%).
La célula que alberga el exogenote abortivo puede expresar los genes de éste: por lo tanto, la célula transductante abortiva contiene proteínas derivadas del exogenote. Parte de las proteínas (en principio la mitad), pasarán a la célula hija que no reciba el exogenote en la siguiente generación. Las hijas de la hija (“las nietas no herederas del original”) reciben la cuarta parte, etc... es decir, aparte de la célula hija que en cada generación hereda el exogenote, sus parientes no-herederos más cercanos reciben proteínas derivadas de la expresión previa de los genes del exogenote. Esto significa que las células no herederas pueden poseer, durante unas pocas generaciones, la función o funciones del exogenote, hasta que el producto correspondiente se diluya o se inactive.
Esto permite detectar fácilmente determinados tipos de transductantes abortivos, distinguiéndolos de los transductantes completos. Por ejemplo, si estamos seleccionando transductantes en base a la adquisición, por parte de una cepa receptora auxotrofa, de la versión silvestre del gen que tiene mutado, sembrando en placas de Petri con medio mínimo, se distinguen dos dos tipos de colonias:
colonias grandes, correspondientes a transductantes completos;
| |
microcolonias, correspondientes a los transductantes abortivos.
|
d) Si el exogenote es un plásmido, al llegar a la célula receptora, puede replicarse autónomamente.
e) Si el exogenote contiene un transposón, éste puede insertarse en el genoma de la célula receptora.
No todos los fagos virulentos pueden actuar como mediadores de transducción generalizada. Los fagos con esta capacidad suelen ser aquellos que no degradan totalmente el ADN del hospedador inmediatamente después de entrar (de otra manera, no habría ADN intacto que transducir). Además, su sistema de empaquetamiento de ADN no debe ser muy específico: fagos como el P22 de Salmonella typhimurium o el P1 de Escherichia coli tienen un mecanismo de empaquetado secuencial de unidades de concatémeros, mecanismo que reconoce secuencias que también existen con cierta frecuencia en el genoma del hospedador.
La transducción generalizada ha sido muy útil en el análisis genético de bacterias y la construcción de nuevas cepas. El alumno seguramente estudiará en la asignatura de Genética algunas aplicaciones, incluida la elaboración de mapas de ligamiento. En las prácticas de Virología del Departamento de Microbiología de la Facultad de Ciencias los estudiantes realizan un interesante experimento de co-transducción que permite aplicar algunas de estas ideas.
TRANSCRIPCIÓN
La traducción es el proceso por el cual un molécula de ARN mensajero se transforma en una secuencia de aminoácidos (proteínas/enzimas).
Iniciación
Hay un único codón que codifica para metionina. Pero hay dos tRNA: tRNAfMet y tRNAMet en los que el primero es el que se usa cuando AUG representa el codón de inicio y el segundo para AUG en posiciones interiores.
La subunidad pequeña ribosómica se fija al factor de iniciación IF3 que impide que las dos subunidades se fijen. Para esto ayuda IF1.
Se fija el mRNA a la subunidad de tal forma que AUG se sigua en el lugar preciso, junto con él se fijan IF2 y GTP. AUG es conducido a la posición correcta en la subunidad gracias a que se reconoce una señal iniciadora (Shine-Dalgarno) que es rica en purinas y se emparejan las bases del rRNA 16 S con esta secuencia.
Así se posiciona correctamente AUG. En eucariotas, la subunidad pequeña se une al casquete y corre hasta el AUG.
Los ribosomas tienen dos sitios el sitio A (aminoacilo) y el sitio P (peptidilo). En P se posiciona AUG que es el único sitio en el que se puede posicionar el f-Met-tRNAfMet. Ahora se libera IF3, y la unidad mayor se acopla hidrolizando GTP y liberando IF1 y IF2.
Elongación
El ciclo de elongación se produce en tres pasos: entrada, enlace peptídico y traslocación. Los factores de elongación catalizan: EF-G la traslocación, EF-TS desplaza GTP de EF-TU y EF-TU forma el complejo aa-tRNA.
Ya tenemos fijada la formilmetionina y el siguiente paso es el primero de la elongación. El segundo aa-tRNA entra fijado a EF-TU que también contiene GTP unido. Este aa-tRNA se une al sitio A del ribosoma cosa que va acompañada de la hidrólisis de GTP y entonces EF-TU-GDP abandona el ribosoma. Se regenera entonces el GDP mediante EF-TS que quita a GDP para hacer hueco a GTP y de nuevo comenzar este ciclo.
A continuación se produce un desplazamiento nucleofílico del tRNA del stio P por el grupo amino de un tRNA situado en A. El aminoácido se transfiere al sitio A y queda el tRNA libre en P, se produce una transpeptidización que cataliza la subunidad grande, el centro activo es peptidil-transferasa.
El tercer paso o translocación consiste en que el ribosoma se traslada un codón hacia el extremo 3' del mRNA utilizando energía proporcionada por la hidrólisis de GTP unido a EF-F. Así se deja el stio A libre y el dipeptidil-tRNA está en P.
Terminación
Para la terminación de las cadenas es fundamental la presencia de los factores de terminación.
RF3 se une a GTP y estimula la unión al ribosoma deRF1 y RF2 que actúan a nivel de A. Entonces se hidroliza el enlace éster entre el polipéptido en crecimiento y el tRNA del sitio P y se libera el polipéptido acabado. Posteriormente se libera el ribosoma, el mRNA y el tRNA desacilado y el factor de liberación RF3.
DESCIFRAMIENTO DEL CÓDIGO GENÉTICO
El aminoácido codificado debe estarlo por un número pequeño pero no uno ni dos porque las combinaciones respectivas darían un máximo de 4 y 16 aa y son 20 los fundamentales. Mediante mutaciones se probó que el código sólo puede ser secuenciado por tripletes de nucleótidos. Es un código no solapado y en el que no hay puntuaciones entre codones. Si el código fuera solapado a la hora de las inserciones y deleciones se producirían problemas graves. Al no ser solapado tenemos tres marcos de lectura posibles, pero sólo uno de ellos es verdadero.
El código genético, además, es degenerado, pues existe cada aminoácido es secuenciado por más de un codón.
En 1961 Marshall Nirenberg y Heninrich Matthaei incubaron un polirribonucleótido de poliU con un extracto de E. coli, GTP y una mezcla de los 20 aminoácidos. en tubos diferentes. En cada tubo marcaron de forma radiactiva un aminoácido distinto. El polipéptido radiactivo se formó en el tubo de Phe y por tanto concluyeron que UUU codificaba Phe. De la misma manera se demostró para CCC y AAA. Con estos métodos podían hallar la composición de bases pero no su secuencia.
Por esta época Khorana aporto otro método que permitía sintetizar polirribonucleótidos con secuencias repetitivas y definidas de dos a cuatro bases.
HIPÓTESIS DEL BALANCEO
Los tRNA reconocen codones medinte apareamiento de bases del codón del mRNA y una secuencia de tres bases del tRNA denominada anticodón.
Los dos RNA se aparean antiparalelamente. La primera base del codón se encuentra en el extremo 5' pues el mRNA va en dirección 5'-3' y la primera del tRNA se encuentra en el extremo 3' ya que va en dirección 3'-5'.
El número de tRNA para cada aminoácido no es el mismo que el número de sus codones Además, algunos de los tRNA tiene inosinato (I), que contiene la base poco frecuente hipoxantina que puede aparearse con U, C y A, aunque son más débiles que los habituales.
Las terceras bases de los codones forman puentes de hidrógeno bastante débiles con el resíduo I del anticodón.
Crick observó que la tercera base de casi todos los codones se aparea de manera bastante suelta con su correspondiente, es decir, la tercera base se balancea.
Las primeras bases de un codón en el mRNA siempre forman pares de bases de Watson y Crick con las bases del anticodón de tRNA confiriéndole así la parte más importante de la especificidad a las dos primeras.
La primera base de un anticodón (leyendo en 5'-3') determina el número de codones leídos por un tRNA dado dada la especificidad mayor o menor del enlace:
- ARNt 321
- ARNm 123
Cuando un aminoácido es especificado por varios codones y éstos difieren en cualquiera de las dos primeras bases, requieren tRNAs diferentes.
Para traducir los 61 codones se necesita un número mínimo de 32 tRNA.
La tercera base es la del balanceo y permite la rápida disociación del tRNA de su codón durante la síntesis.
El código genético es casi universal difiriendo en aquellos organismos que se diversificaron muy pronto y en las mitocondrias.

.jpg)


No hay comentarios:
Publicar un comentario